O Google Deepmind retirou uma nova versão do AlphaFold, seu modelo transformador de aprendizado de máquina que prevê a forma e o comportamento das proteínas. AlphaFold 3 não é apenas mais preciso, mas prevê interações com outras biomoléculas, tornando-o uma ferramenta de pesquisa muito mais versátil – e a empresa está disponibilizando uma versão limitada do modelo gratuitamente para uso online.
Desde a estreia do primeiro AlphaFold em 2018, o modelo continua sendo o principal método de previsão da estrutura das proteínas a partir da sequência de aminoácidos que as compõem.
Embora isso pareça uma tarefa bastante limitada, é fundamental para quase toda a biologia compreender as proteínas – que realizam uma variedade quase infinita de tarefas em nossos corpos – no nível molecular. Nos últimos anos, técnicas de modelagem computacional como AlphaFold e RoseTTaFold substituíram métodos caros baseados em laboratório, acelerando o trabalho de milhares de pesquisadores em vários campos.
Mas a tecnologia ainda é um trabalho em andamento, com cada modelo “apenas um passo no caminho”, como disse o fundador da Deepmind, Demis Hassabis, em uma teleconferência sobre o novo sistema. A empresa provocou o lançamento no final do ano passado, mas isso marca sua estreia oficial.
Deixarei os blogs científicos explicarem exatamente como o novo modelo melhora os resultados, mas basta dizer aqui que uma variedade de melhorias e técnicas de modelagem tornaram o AlphaFold 3 não apenas mais preciso, mas também mais amplamente aplicável.
Uma das limitações da modelagem de proteínas é que mesmo que você saiba qual será a forma que uma sequência de aminoácidos assumirá, isso não significa que você necessariamente sabe a quais outras moléculas ela se ligará e como. E se você quiser realmente fazer coisas com essas moléculas, o que a maioria faz, você precisa descobrir isso por meio de modelagem e testes mais trabalhosos.
“A biologia é um sistema dinâmico, e você tem que entender como as propriedades da biologia surgiram através da interações entre diferentes moléculas na célula. E você pode pensar no AlphaFold 3 como nosso primeiro grande passo nessa direção”, disse Hassabis. “É capaz de modelar proteínas interagindo, é claro, com outras proteínas, mas também com outras biomoléculas, incluindo, principalmente, cadeias de DNA e RNA.”
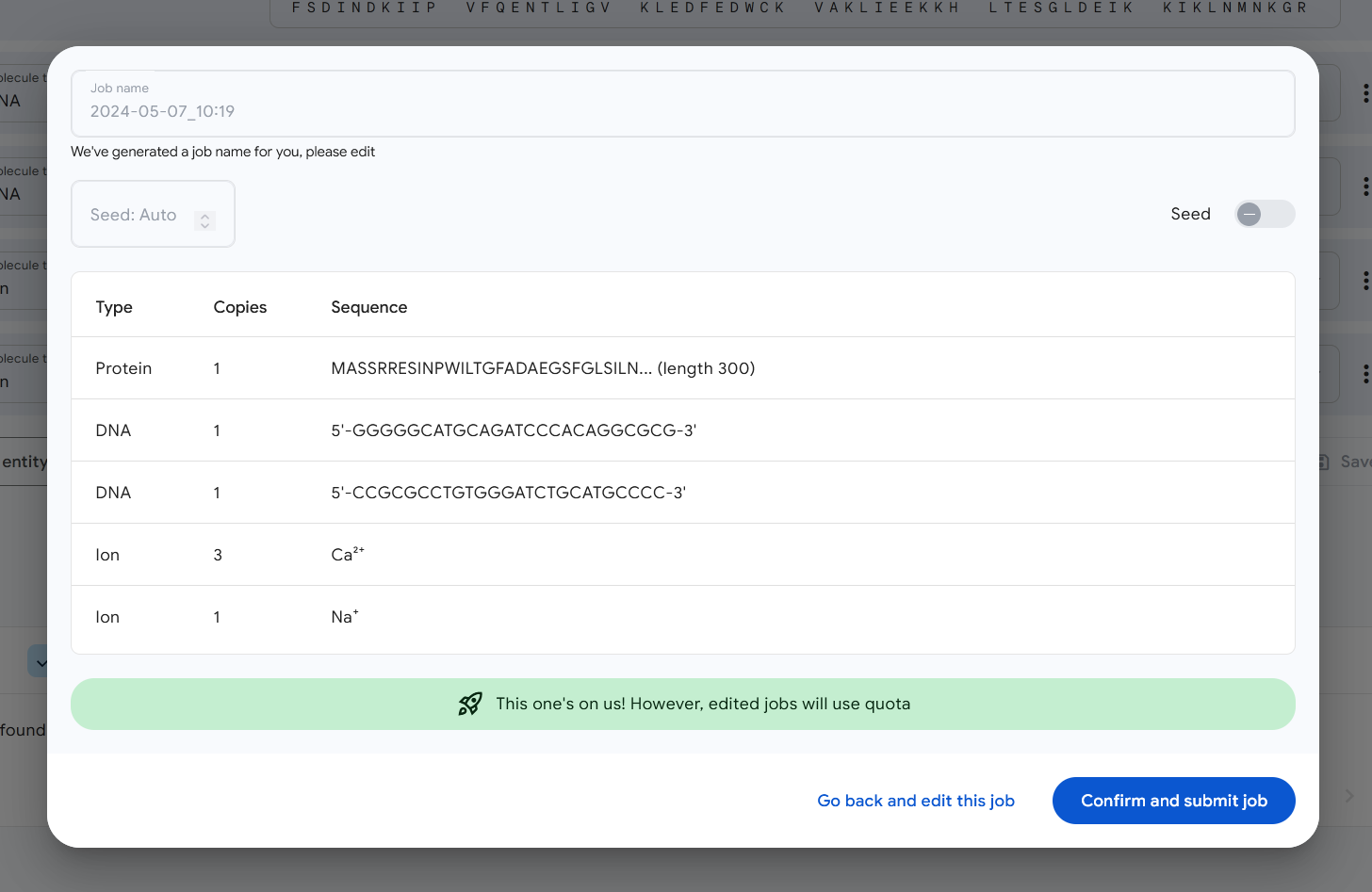
O AlphaFold 3 permite simular múltiplas moléculas ao mesmo tempo – por exemplo, uma cadeia de DNA, algumas moléculas de ligação ao DNA e talvez alguns íons para apimentar as coisas. Aqui está o que você obtém com uma combinação específica, com as fitas de DNA subindo no meio, as proteínas aglomeradas nas laterais, e acho que esses são os íons aninhados no meio, como pequenos ovos:
Isto, claro, não é uma descoberta científica em si. Mas mesmo descobrir se uma proteína experimental se ligaria de alguma forma, ou desta forma, ou se contorceria para obter esta forma, era geralmente o trabalho de dias, pelo menos, ou talvez de semanas a meses.
Embora seja difícil exagerar o entusiasmo neste campo nos últimos anos, os pesquisadores têm sido em grande parte prejudicados pela falta de modelagem de interação (da qual a nova versão oferece uma forma) e pela dificuldade de implantar o modelo.
Esta segunda questão é talvez a maior das duas, pois embora as novas técnicas de modelação fossem “abertas” em certo sentido, tal como outros modelos de IA, não são necessariamente simples de implementar e operar. É por isso que o Google Deepmind está oferecendo o AlphaFold Server, um aplicativo da web gratuito e totalmente hospedado que disponibiliza o modelo para uso não comercial.
É gratuito e muito fácil de usar – fiz isso em outra janela da chamada enquanto eles explicavam (foi assim que consegui a imagem acima). Você só precisa de uma conta do Google e, em seguida, alimente-a com tantas sequências e categorias quanto ela puder suportar – há alguns exemplos fornecidos – e envie; em alguns minutos seu trabalho estará concluído e você receberá uma molécula 3D viva colorida para representar a confiança do modelo na conformação naquela posição. Como você pode ver acima, as pontas das fitas e as partes mais expostas a átomos rebeldes são mais claras ou vermelhas para indicar menos confiança.
Perguntei se havia alguma diferença real entre o modelo disponível publicamente e o utilizado internamente; Hassabis disse que “disponibilizamos a maioria das capacidades do novo modelo”, mas não deu mais detalhes.
É claramente o Google jogando seu peso – enquanto, até certo ponto, mantém as melhores partes para si, o que, claro, é sua prerrogativa. Criar uma ferramenta hospedada e gratuita como essa envolve dedicar recursos consideráveis à tarefa — não se engane, isso é um poço de dinheiro, uma versão shareware cara (para o Google) para convencer os pesquisadores de todo o mundo de que o AlphaFold 3 deveria ser, no mínimo pelo menos, uma flecha em sua aljava.
Créditos da imagem: Google Deep Mind
Tudo bem, porém, porque a tecnologia provavelmente imprimirá dinheiro por meio da subsidiária Alphabet (o que a torna prima do Google?) Isomórfica Labs, que está colocando ferramentas computacionais como AlphaFold para trabalhar no design de medicamentos. Bem, todo mundo está usando ferramentas computacionais hoje em dia – mas o Isomorphic foi o primeiro a explorar os modelos mais recentes do Deepmind, combinando-os com “algumas coisas mais proprietárias relacionadas à descoberta de medicamentos”, como observou Hassabis. A empresa já possui parcerias com Eli Lilly e Novartis.
No entanto, AlphaFold não é o princípio e o fim de tudo da biologia – apenas uma ferramenta muito útil, como inúmeros pesquisadores concordarão. E permite-lhes fazer o que Max Jaderberg da Isomórfica chamou de “design racional de medicamentos”.
“Se pensarmos, no dia a dia, como isso tem impacto nos laboratórios isomórficos: permite que nossos cientistas, nossos projetistas de medicamentos, criem e testem hipóteses em nível atômico e, em segundos, produzam previsões de estrutura altamente precisas… para ajudar os cientistas raciocinam sobre quais são as interações a serem feitas e como avançar nesses projetos para criar um bom medicamento”, disse ele. “Isso é comparado aos meses ou mesmo anos que pode levar para fazer isso experimentalmente.”
Embora muitos comemorem a conquista e a ampla disponibilidade de uma ferramenta hospedada e gratuita como o AlphaFold Server, outros podem apontar, com razão, que isso não é realmente uma vitória para a ciência aberta.
Como muitos modelos proprietários de IA, o processo de treinamento do AlphaFold e outras informações cruciais para replicá-lo – uma parte fundamental do método científico, você deve se lembrar – são em grande parte e cada vez mais retidos. Embora o artigo publicado na Nature aborde os métodos de sua criação com algum detalhe, faltam muitos detalhes e dados importantes, o que significa que os cientistas que desejam usar a ferramenta de biologia molecular mais poderosa do planeta terão que fazê-lo sob o olhar atento da Alphabet, Google e Deepmind (quem sabe quem realmente detém as rédeas).
Os defensores da ciência aberta afirmam há anos que, embora estes avanços sejam notáveis, é sempre melhor, a longo prazo, partilhar abertamente este tipo de coisas. Afinal, é assim que a ciência avança e, na verdade, também é assim que alguns dos softwares mais importantes do mundo evoluíram.
Tornar o AlphaFold Server gratuito para qualquer aplicação acadêmica ou não comercial é, em muitos aspectos, um ato muito generoso. Mas a generosidade do Google raramente vem sem compromisso. Não há dúvida de que muitos pesquisadores aproveitarão esse período de lua de mel para usar o modelo tanto quanto for humanamente possível, antes que o outro sapato caia.





